🔍【Nat. Biomed. Eng】19个基础模型横评:数字病理AI的性能大比拼!
文章信息
📚文章标题:Benchmarking foundation models as feature extractors for weakly supervised computational pathology 作者:Jakob Nikolas Kather 等 期刊:Nature Biomedical Engineering 链接:https://doi.org/10.1038/s41551-025-01516-3
近年来,基础模型(Foundation Models)在计算病理学领域掀起了革命性的浪潮。它们通过自监督学习在大规模组织切片图像上训练,能够提取丰富的图像特征,进而实现癌症分型、预后预测、分子标志物识别等多种临床任务。然而,尽管基础模型层出不穷,真正系统性、独立性的评估研究却寥寥无几。为此,Nature Biomedical Engineering 于2025年10月发表了一项重磅研究,对19个主流病理基础模型在13个独立患者队列、共计9,528张切片图像上的31项临床相关任务中进行了全面评估。
🧪研究设计:19个模型 × 13个队列 × 31项任务
本研究聚焦于肺癌、结直肠癌、胃癌和乳腺癌四大癌种,涵盖6,818名患者的9,528张H&E染色的全切片图像(WSIs)。研究团队从多个国家的真实临床数据中构建了13个独立队列,确保训练与测试数据完全隔离,避免数据泄漏。
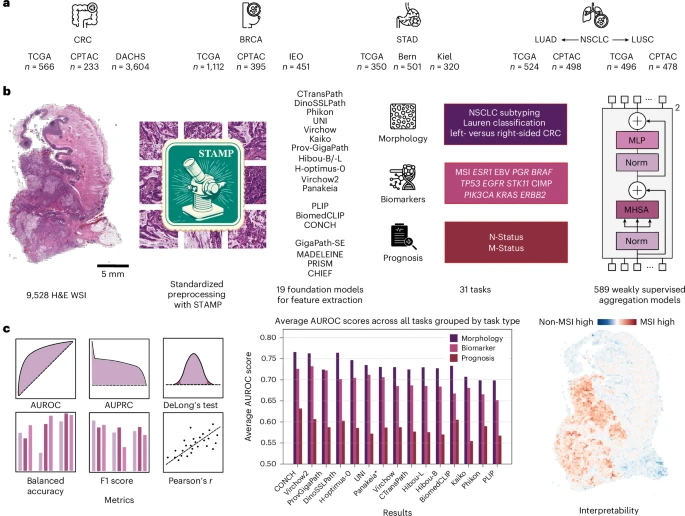
所评估的19个基础模型包括:
- 12个视觉模型(如Virchow2、DinoSSLPath、CTransPath等)
- 3个视觉-语言模型(如CONCH、BiomedCLIP、PLIP)
- 4个幻灯片编码器(如PRISM、CHIEF等)
这些模型被应用于31项弱监督任务,包括:
- 5项形态学分类任务
- 19项生物标志物预测任务
- 7项预后相关任务
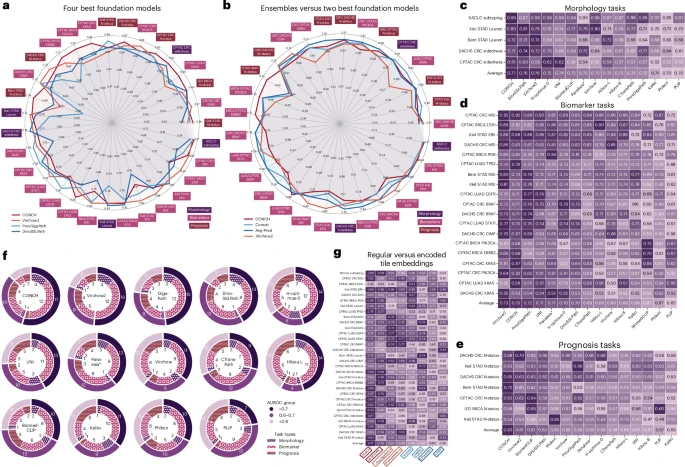
🏆性能排名:CONCH与Virchow2领跑群雄
研究采用AUROC(受试者工作特征曲线下面积)作为主要评估指标,结果显示:
- 🔹 在所有任务中,CONCH和Virchow2平均AUROC均为0.71,表现最优。
- 🔹 在形态学任务中,CONCH以0.77的平均AUROC领先,Virchow2紧随其后(0.76)。
- 🔹 在生物标志物预测中,Virchow2与CONCH并列第一(AUROC均为0.73)。
- 🔹 在预后任务中,CONCH再次领先(AUROC为0.63),Virchow2和BiomedCLIP紧随其后(0.61)。
此外,CONCH在精确率-召回曲线(AUPRC)、F1分数和平衡准确率等指标上也表现最佳。
📉数据稀缺场景下的挑战与机遇
基础模型的一个重要优势在于其在小样本场景下的泛化能力。研究进一步在75、150和300名患者的子集上训练下游模型,模拟真实临床中常见的低样本情境。结果发现:
- 在300人样本中,Virchow2在8项任务中表现最佳。
- 在150人样本中,PRISM领先(9项任务),Virchow2次之(6项)。
- 在75人样本中,CONCH表现突出(5项任务),显示其在极低样本下的鲁棒性。
然而,整体来看,所有模型在样本减少时性能均有所下降,说明当前基础模型在极端数据稀缺场景下仍存在局限。 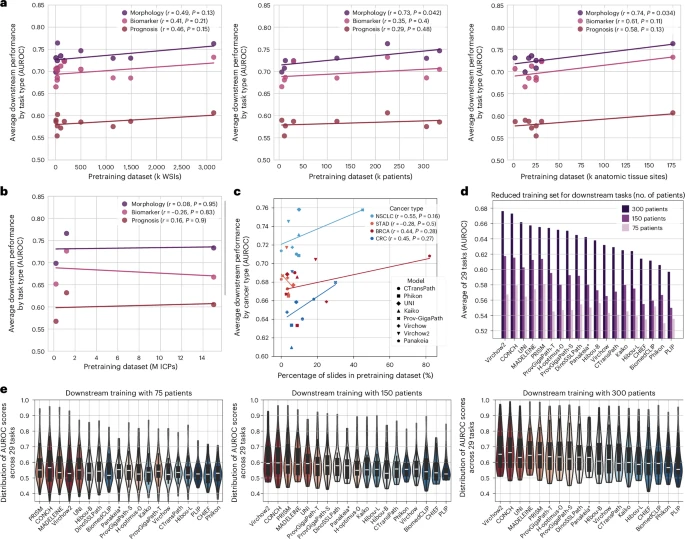
🧬数据多样性胜于数据量
一个重要发现是:训练数据的“多样性”比“数量”更能提升模型性能。研究发现:
- 组织类型的多样性与模型在形态学任务中的表现呈显著正相关(r=0.74)。
- Virchow2虽然训练数据量(310万张WSIs)远超CONCH(117万图文对),但两者性能相当,说明数据质量与多样性同样关键。
- Panakeia模型虽仅在乳腺癌和结直肠癌上训练,但在胃癌和肺癌任务中也表现不俗,显示出一定的跨癌种泛化能力。
🔍模型关注区域差异显著,融合策略提升性能
通过热力图分析,研究发现不同模型在预测同一任务时关注的组织区域存在显著差异:
- 高性能模型(如CONCH、Virchow2)更倾向于聚焦于肿瘤区域。
- 某些模型(如Kaiko、Hibou)则可能错误关注笔迹等无关区域,提示其可能存在“伪相关”问题。
- Cohen's kappa分析显示模型间预测一致性中等,说明它们学习到的特征具有互补性。
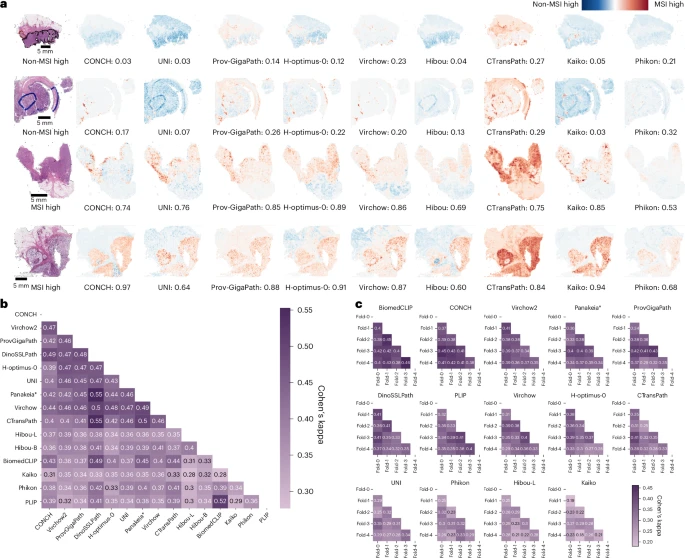
基于此,研究尝试了两种模型融合策略:
- 平均多个模型的预测分数
- 拼接多个模型的特征向量
结果显示,融合模型在55%的任务中优于单一模型,平均AUROC提升1.2%,错误分类率下降6.2%。其中,CONCH与Virchow2的融合模型在29项任务中有9项显著优于单独模型。
🧠关键启示:基础模型的未来发展方向
这项研究不仅提供了详尽的模型性能对比,更提出了多个关键建议:
- 📌 多模态训练(图像+文本)有助于提升图像特征表达能力,即使最终应用仅基于图像。
- 📌 数据多样性(组织类型、种族、癌种)比单纯增加图像数量更能提升模型泛化能力。
- 📌 局部形态特征在多数任务中足以支持准确预测,提示未来模型可优化为更高效的tile级别编码器。
- 📌 当前模型在低阳性率任务(如EGFR突变、MSI状态)中仍有改进空间,需进一步优化架构与训练策略。
🧩结语:为数字病理AI发展指明方向
这项涵盖19个基础模型、31项任务、13个独立队列的研究,为数字病理AI模型的选择与开发提供了宝贵的参考。它不仅揭示了当前模型的性能天花板,也指出了未来优化的关键路径——数据多样性、多模态训练与模型融合。对于希望在临床实践中部署AI病理工具的研究者与机构而言,这无疑是一份具有里程碑意义的指南。